 Эта статья посвящена языку R. Он не так широко распространен на территории ex-USSR, как Matlab и тем более Python, но, безусловно, заслуживает внимания. Нельзя не отметить, что R — фактически стандарт для Data Science (хотя тут хорошо написано, что не R единым живут data scientists). Богатый синтаксис, совместимость с legacy кодом (что весьма важно в научных приложениях), удобная среда разработки RStudio и наличие огромного числа библиотек в CRAN делают R таковым.
Эта статья посвящена языку R. Он не так широко распространен на территории ex-USSR, как Matlab и тем более Python, но, безусловно, заслуживает внимания. Нельзя не отметить, что R — фактически стандарт для Data Science (хотя тут хорошо написано, что не R единым живут data scientists). Богатый синтаксис, совместимость с legacy кодом (что весьма важно в научных приложениях), удобная среда разработки RStudio и наличие огромного числа библиотек в CRAN делают R таковым. Но, как и всегда, оборотной стороной медали (или вернее сказать — интерпретатора R) является скорость вычислений. R поддерживает векторизованные вычисления — семейство функций apply позволяет избегать использования циклов. Но это, в общем-то, синтаксический сахар, и зачастую эффект «ускорения» от использования *apply прямо таки отрицательный. На этом фоне вполне логичным шагом (если не рассматривать переход на языки более низкого уровня) выглядит распараллеливание вычислений. Не секрет, что код поддается этой процедуре с трудом.
Для дальнейшей работы потребуются два пакета — «foreach» (позволяет использовать конструкцию foreach) и «doSNOW» (обеспечивает поддержку параллельных вычислений). Из CRAN их можно установить так:
install.packages(c("foreach","doSNOW"))Следующие команды — подготовка «кластера» из трех ядер процессора к работе:
library(foreach) library(doSNOW) cluster <- makeCluster(3, type = "SOCK", outfile="") registerDoSNOW(cluster) Рассмотрим пример, где подсчитывается сумма квадратов чисел от 1 до 10000:
system.time ({ sum.par <- foreach(i=1:10000, .combine='+') %dopar% { i^2 }}) В принципе, все очень похоже на обычный цикл, за исключением того, что надо указывать, как группировать результаты — в данном случае это операция сложения (а часто это функции c, rbind, cbind — или же собственноручно написанная функция). Если не важно, в каком порядке группировать, то использование аргумента .inorder=F позволяет несколько ускорить вычисления. Если вместо %dopar% использовать директиву %do%, то вычисления будут выполняться последовательно (т.е. как в обычном цикле). К слову сказать, для такой простой задачи время выполнения в последовательном варианте будет заметно меньше, т.к. накладные раcходы на манипуляции с данными в параллельном варианте занимают далеко не самое последнее место в балансе.
Перейдем к более сложной задаче. Допустим, у нас есть вектор случайных величин х.
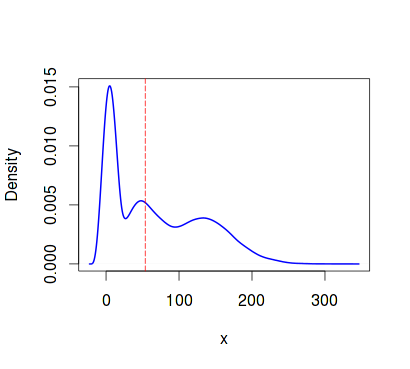
trials <- 10000 n <- 24000 alfa <- 1.6 beta <- 3 set.seed(1) x <- c(rgamma(n/3, shape=alfa, scale=beta), rgamma(n/3, shape=3*alfa, scale=4*beta), rgamma(n/3, shape=10*alfa, scale=3*beta)) plot(density(x, kernel="cosine"),lwd=2, col="blue", xlab="x",ylab="Density", main="Density plot") abline(v = median(x), col = "red", lty=5)
Оценим медиану — с указанием доверительных интервалов. Для этого применим бутстрэп и измерим время выполнения для последовательного кода и параллельного соответственно:
set.seed(1) seq.time <- system.time({ seq.medians <- foreach(icount(trials), .combine=cbind) %do% { median(sample(x, replace = T)) } })
> quantile(seq.medians, c(.025, .975)) 2.5% 97.5% 52.56311 54.99636 > seq.time user system elapsed 19.884 0.252 20.138
set.seed(1) par.time <- system.time({ par.medians <- foreach(icount(trials), .combine=cbind) %dopar% { median(sample(x, replace = T)) } })
> quantile(par.medians, c(.025, .975)) 2.5% 97.5% 52.56257 54.94890 > par.time user system elapsed 4.252 0.420 10.893 Разница вполне ощутима. Оценим время работы последовательного «векторизированного» варианта для этой же задачи (с применением функции apply):
set.seed(1) seq.time.ap <- system.time({ seq.medians.ap <- apply(matrix(sample(x, trials*n, replace = T), trials, n), 1, median) })
> quantile(seq.medians.ap, c(.025, .975)) 2.5% 97.5% 52.56284 54.98266 > seq.time.ap user system elapsed 23.732 1.728 25.472 Подобный же подход можно использовать для определения параметров алгоритмов машинного обучения. Например, в пакете «RSNNS» есть функции для обучения сети Элмана (насколько я знаю, в R, в общем-то, нет других пакетов для работы с рекуррентными сетями). Выбор количества нейронов и скорости обучения — довольно ресурсоемкая задача, посему есть смысл в распараллеливании.
Перейдем от сферических примеров к более насущной проблеме. Для этого установим два пакета:
install.packages(c("ElemStatLearn","RSNNS")) Из пакета «ElemStatLearn» нам понадобятся два набора данных — zip.train и zip.test, которые содержат сжатые (gzipped) образы рукописных цифр. Датасеты довольно объемные, поэтому возьмем лишь их часть: соответственно для обучения сети, валидации и тестирования.
library("ElemStatLearn") library(RSNNS) data(zip.train) data(zip.test) image(zip2image(zip.train, 1000), col=gray(255:0/255)) train <- zip.train[1:1000,-1] trainC <- decodeClassLabels(zip.train[1:1000,1]) valid <- zip.train[1001:1200,-1] validC <- zip.train[1001:1200,1] test <- zip.test[1:1000,-1] testC <- zip.test[1:1000,1]
Прежде чем обучать несколько сетей параллельно, нам надо продумать, что именно мы хотим получить. Допустим, нас интересуют два параметра нейронной сети — количество нейронов в скрытом слое и скорость обучения, и мы хотим подобрать более-менее оптимальные их значения. Значит, результатом работы будет кадр данных, содержащий три числа — количество нейронов, скорость обучения и ошибку, на основе которой мы и будем определять оптимальность параметров. Ранее я упоминал о функции, которая осуществляет группировку результатов работы параллельного цикла. В случае данных, которые имеют более сложную структуру, чем у вектора, такую функцию придется писать самому.
comb <- function(df1, df2) if (df1$err < df2$err) df1 else df2 Как видно из кода, функция сравнивает поле err двух кадров данных и выбирает тот, где ошибка меньше. Ниже непосредственно представлен фрагмент скрипта, который и осуществляет обучение сетей в параллельном режиме. Запускаем его и некоторое время любуемся 100% загрузкой трех ядер.
errCl <- function (predicted, true){ return(round(sum(predicted != true)/length(true), 4)) } size <- c(13,29,53,71) lr <- c(.001,.01,.1,.5) nn.time <- system.time({ elm_par <- foreach(s=size, .combine='comb') %:% foreach(r=lr, .packages='RSNNS', .combine='comb', .inorder=F) %dopar% { elm <- elman(x=train, y=trainC, size=s, learnFunc="JE_BP", learnFuncParams=r, linOut=F, maxit=193) pred <- max.col(predict(elm, valid)) - 1 e <- errCl(pred, validC) data.frame(opt_size=s, lrate=r, err=e) } }) Функция errCl используется для определения ошибки классификации. В векторах size и lr содержатся соответственно количества нейронов в скрытом слое сети и скорости обучения сети. Обратите внимание, как организовывается вложенный цикл с помощью инструкции %:%. Собственно, если не вдаваться в подробности, в теле цикла происходит то, что обычно делают при выборе параметров: обучается модель на тренировочном наборе данных, потом используется валидирующий набор данных, определяется ошибка классификации и выбираются те параметры, при которых ошибка меньше. У меня же получился такой результат:
> elm_par opt_size lrate err 1 29 0.5 0.05 > nn.time user system elapsed 0.312 0.168 179.542 Хотя, конечно, не стоит обольщаться ошибкой в 5%. Проверим нашу сеть с 29 нейронами и скоростью обучения 0.5 на тестовом наборе:
elm <- elman(x=train, y=trainC, size=29, learnFunc="JE_BP", learnFuncParams=0.5, linOut=F, maxit=193) pred <- max.col(predict(elm, test)) - 1 e <- errCl(pred, testC)
> e [1] 0.119 Что же, 11.9% тоже весьма неплохо. Теперь дело осталось за малым — «погасить» кластер
stopCluster(cluster)
Вместо заключения
R — весьма удобный и коварный язык. Привыкнув к нему — и особенно к разнообразию пакетов в CRAN — трудно перейти на что-то другое. Но, безусловно, у каждого языка своя ниша. Для R — это статистика и анализ данных. И хотя R может быть весьма неторопливым, для многих задач его производительности вполне хватает.
Литература
High-Performance and Parallel Computing with R
Nesting Foreach Loops
RSNNS Package
ссылка на оригинал статьи http://habrahabr.ru/post/206892/
Добавить комментарий