Привет, хабр!
Как и обещал, продолжаю публикацию разборов задач, которые я прорешал за время работы с ребятами из MLClass.ru. В этот раз мы разберем метод главных компонент на примере известной задачи распознавания цифр Digit Recognizer с платформы Kaggle. Статья будет полезна новичкам, которые еще только начинают изучать анализ данных. Кстати, еще не поздно записаться на курс Прикладной анализ данных, получив возможность максимально быстро прокачаться в данной области.
Вступление
Данная работа является естественным продолжением исследования, изучающего зависимость качества модели от размера выборки. В ней для была показана возможность уменьшения количества используемых объектов в обучающей выборке с целью получения приемлемых результатов в условиях ограниченных вычислительных ресурсов. Но, кроме количества объектов, на размер данных влияет и количество используемых признаков. Рассмотрим эту возможность на тех же данных. Используемые данные были подробно изучены в предыдущей работе, поэтому просто загрузим тренировочную выборку в R.
library(readr) library(caret) library(ggbiplot) library(ggplot2) library(dplyr) library(rgl) data_train <- read_csv("train.csv") ## |================================================================================| 100% 73 MB Как мы уже знаем данные имеют 42000 объектов и 784 признака, представляющие собой значение яркости каждого из пикселей составляющего изображение цифры. Разобъём выборку на тренировочную и тестовую в соотношении 60/40.
set.seed(111) split <- createDataPartition(data_train$label, p = 0.6, list = FALSE) train <- slice(data_train, split) test <- slice(data_train, -split) Теперь удалим признаки, имеющие константное значение.
zero_var_col <- nearZeroVar(train, saveMetrics = T) train <- train[, !zero_var_col$nzv] test <- test[, !zero_var_col$nzv] dim(train) ## [1] 25201 253 В итоге осталось 253 признака.
Теория
Метод главных компонент (PCA) преобразует базовые признаки в новые, каждый из которых является линейной комбинацией изначальных таким образом, что разброс данных (то есть среднеквадратичное отклонение от среднего значения) вдоль них максимален. Метод применяется для визуализации данных и для уменьшения размерности данных (сжатия).
PCA
Для большей наглядности случайным образом отберём из тренировочной выборки 1000 объектов и изобразим их в пространстве первых двух признаков.
train_1000 <- train[sample(nrow(train), size = 1000),] ggplot(data = train_1000, aes(x = pixel152, y = pixel153, color = factor(label))) + geom_point() 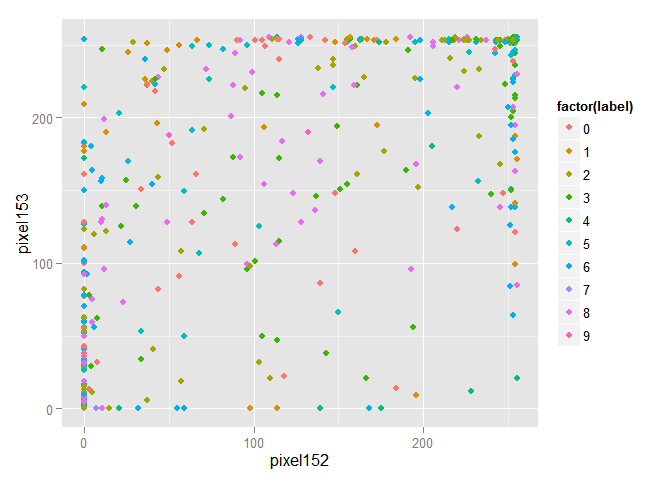
Очевидно, что объекты перемешаны и выделить среди них группы объектов принадлежащих одному классу проблематично. Проведём преобразование данных по методу главных компонент и изобразим в пространстве первых двух компонент. Замечу, что компоненты расположены в убывающем порядке в зависимости от разброса данных, который приходится вдоль них.
pc <- princomp(train_1000[, -1], cor=TRUE, scores=TRUE) ggbiplot(pc, obs.scale = 1, var.scale = 1, groups = factor(train_1000$label), ellipse = TRUE, circle = F, var.axes = F) + scale_color_discrete(name = '') + theme(legend.direction = 'horizontal', legend.position = 'top') 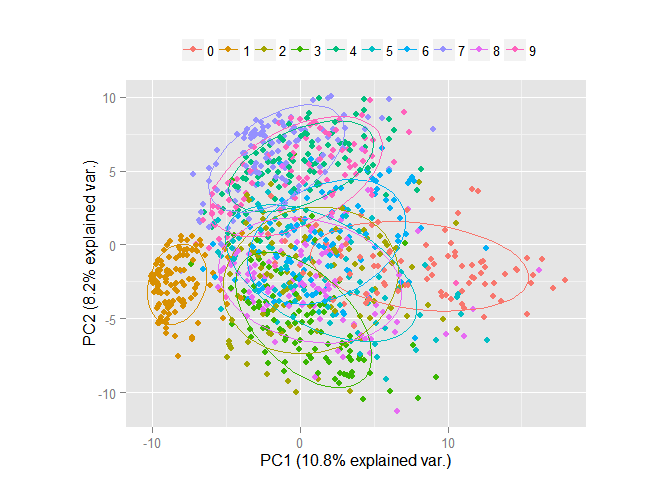
Очевидно, что даже в пространстве всего лишь двух признаков уже можно выделить явные группы объектов. Теперь рассмотрим те же данные, но уже в пространстве первых трёх компонент.
plot3d(pc$scores[,1:3], col= train_1000$label + 1, size = 0.7, type = "s") 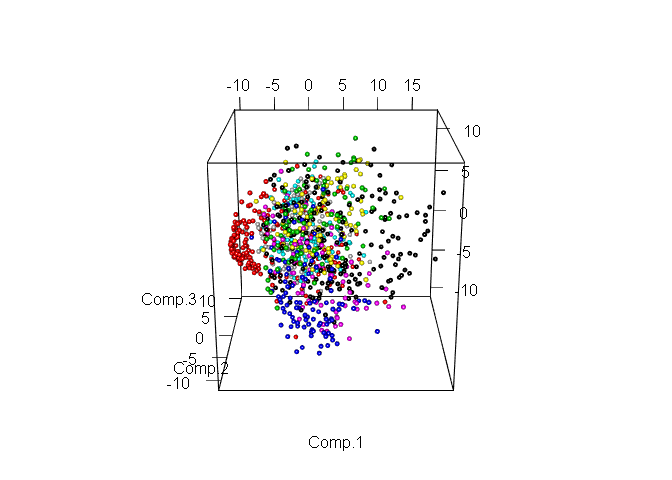
Выделение различных классов ещё больше упростилось. Теперь выберем количество компонент, которое будем использовать для дальнейшей работы. Для этого посмотрим на соотношение дисперсии и количество компонент объясняющие её, но уже используя всю тренировочную выборку.
pc <- princomp(train[, -1], cor=TRUE, scores=TRUE) variance <- pc$sdev^2/sum(pc$sdev^2) cumvar <- cumsum(variance) cumvar <- data.frame(PC = 1:252, CumVar = cumvar) ggplot(data = cumvar, aes(x = PC, y = CumVar)) + geom_point() 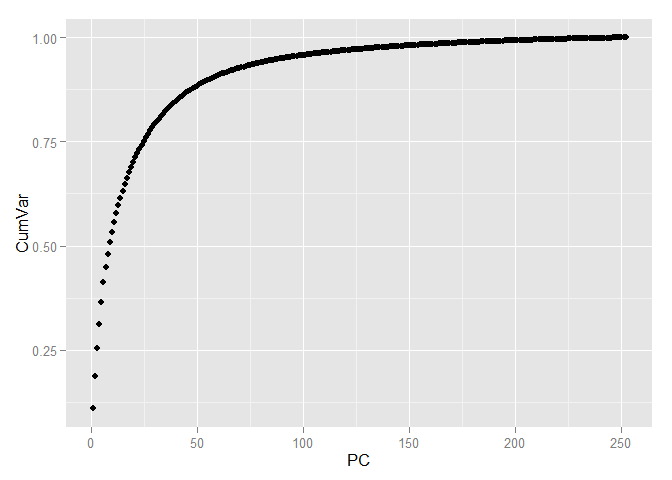
variance <- data.frame(PC = 1:252, Var = variance*100) ggplot(data = variance[1:10,], aes(x = factor(PC), y = Var)) + geom_bar(stat = "identity") 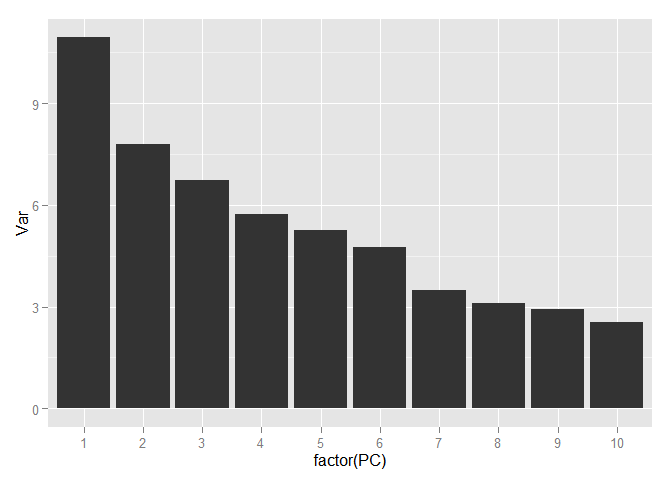
sum(variance$Var[1:70]) ## [1] 92.69142 Для того, чтобы сохранить более 90 процентов информации, содержащейся в данных достаточно всего лишь 70 компонент, т.е. мы от 784 признаков пришли к 70 и, при этом, потеряли менее 10 процентов вариации данных!
Преобразуем тренировочную и тестовую выборки в пространство главных компонент.
train <- predict(pc) %>% cbind(train$label, .) %>% as.data.frame(.) %>% select(1:71) colnames(train)[1]<- "label" train$label <- as.factor(train$label) test %<>% predict(pc, .) %>% cbind(test$label, .) %>% as.data.frame(.) %>% select(1:71) colnames(test)[1]<- "label" Для выбора параметров моделей я использую пакет caret, предоставляющий возможность выполнять параллельные вычисления, используя многоядерность современных процессоров.
library("doParallel") cl <- makePSOCKcluster(2) registerDoParallel(cl)
KNN
Теперь приступим к созданию предсказывающих моделей используя преобразованные данные. Создадим первую модель используя метод k ближайших соседей (KNN). В этой модели есть только один параметр — количество ближайших объектов, используемых для классификации объекта. Подбирать этот параметр будем с помощью десятикратной кросс-проверки (10-fold cross-validation (CV)) с разбиением выборки на 10 частей. Оценка производится на случайно отобранной части изначальных объектов. Для оценки качества моделей будем использовать метрику Accuracy, представляющий собой процент точно предсказанных классов объектов.
set.seed(111) train_1000 <- train[sample(nrow(train), size = 1000),] ctrl <- trainControl(method="repeatedcv",repeats = 3) Для начала определим область поиска значений параметра. knnFit <- train(label ~ ., data = train_1000, method = "knn", trControl = ctrl,tuneLength = 20) knnFit ## k-Nearest Neighbors ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 899, 901, 900, 901, 899, 899, ... ## Resampling results across tuning parameters: ## ## k Accuracy Kappa Accuracy SD Kappa SD ## 5 0.8749889 0.8608767 0.03637257 0.04047629 ## 7 0.8679743 0.8530101 0.03458659 0.03853048 ## 9 0.8652707 0.8500155 0.03336461 0.03713965 ## 11 0.8529954 0.8363199 0.03692823 0.04114777 ## 13 0.8433141 0.8255274 0.03184725 0.03548771 ## 15 0.8426833 0.8248052 0.04097424 0.04568565 ## 17 0.8423694 0.8244683 0.04070299 0.04540152 ## 19 0.8340150 0.8151256 0.04291349 0.04788273 ## 21 0.8263450 0.8065723 0.03914363 0.04369889 ## 23 0.8200042 0.7995067 0.03872017 0.04320466 ## 25 0.8156764 0.7946582 0.03825163 0.04269085 ## 27 0.8093227 0.7875839 0.04299301 0.04799252 ## 29 0.8010018 0.7783100 0.04252630 0.04747852 ## 31 0.8019849 0.7794036 0.04327120 0.04827493 ## 33 0.7963572 0.7731147 0.04418378 0.04930341 ## 35 0.7936906 0.7701616 0.04012802 0.04478789 ## 37 0.7889930 0.7649252 0.04163075 0.04644193 ## 39 0.7863463 0.7619669 0.03947693 0.04404655 ## 41 0.7829758 0.7582087 0.03482612 0.03889550 ## 43 0.7796388 0.7544879 0.03745359 0.04179976 ## ## Accuracy was used to select the optimal model using the largest value. ## The final value used for the model was k = 5. Теперь сократим её и получим точное значение.
grid <- expand.grid(k=2:5) knnFit <- train(label ~ ., data = train_1000, method = "knn", trControl = ctrl, tuneGrid=grid) knnFit ## k-Nearest Neighbors ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 900, 901, 901, 899, 901, 899, ... ## Resampling results across tuning parameters: ## ## k Accuracy Kappa Accuracy SD Kappa SD ## 2 0.8699952 0.8553199 0.03055544 0.03402108 ## 3 0.8799832 0.8664399 0.02768544 0.03082014 ## 4 0.8736591 0.8593777 0.02591618 0.02888557 ## 5 0.8726753 0.8582703 0.02414173 0.02689738 ## ## Accuracy was used to select the optimal model using the largest value. ## The final value used for the model was k = 3. Наилучший показатель модель имеет при значении параметра k равному 3. Используя это значение получим предсказание на тестовых данных. Построим Confusion Table и вычислим Accuracy.
library(class) prediction_knn <- knn(train, test, train$label, k=3) table(test$label, prediction_knn) ## prediction_knn ## 0 1 2 3 4 5 6 7 8 9 ## 0 1643 0 6 1 0 1 2 0 0 0 ## 1 0 1861 4 1 2 0 0 0 0 0 ## 2 7 7 1647 3 0 0 1 11 0 0 ## 3 1 0 9 1708 2 19 4 6 1 3 ## 4 0 4 0 0 1589 0 10 7 0 6 ## 5 3 2 1 20 1 1474 13 0 6 2 ## 6 0 0 0 1 2 3 1660 0 0 0 ## 7 0 6 3 0 2 0 0 1721 0 13 ## 8 0 1 0 11 1 16 12 4 1522 20 ## 9 0 0 1 3 3 5 1 23 5 1672 sum(diag(table(test$label, prediction_knn)))/nrow(test) ## [1] 0.9820227 Random Forest
Вторая модель — это Random Forest. У этой модели будем выбирать параметр mtry — количество используемых признаков при получении каждого из используемых в ансамбле деревьев. Для выбора наилучшего значения данного параметра пойдём тем же путём, что и ранее.
rfFit <- train(label ~ ., data = train_1000, method = "rf", trControl = ctrl,tuneLength = 3) rfFit ## Random Forest ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 901, 900, 900, 899, 902, 899, ... ## Resampling results across tuning parameters: ## ## mtry Accuracy Kappa Accuracy SD Kappa SD ## 2 0.8526986 0.8358081 0.02889351 0.03226317 ## 36 0.8324051 0.8133909 0.03442843 0.03836844 ## 70 0.8026823 0.7802912 0.03696172 0.04118363 ## ## Accuracy was used to select the optimal model using the largest value. ## The final value used for the model was mtry = 2. grid <- expand.grid(mtry=2:6) rfFit <- train(label ~ ., data = train_1000, method = "rf", trControl = ctrl,tuneGrid=grid) rfFit ## Random Forest ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 898, 900, 900, 901, 900, 898, ... ## Resampling results across tuning parameters: ## ## mtry Accuracy Kappa Accuracy SD Kappa SD ## 2 0.8553016 0.8387134 0.03556811 0.03967709 ## 3 0.8615798 0.8457973 0.03102887 0.03458732 ## 4 0.8669329 0.8517297 0.03306870 0.03690844 ## 5 0.8739532 0.8595897 0.02957395 0.03296439 ## 6 0.8696883 0.8548470 0.03203166 0.03568138 ## ## Accuracy was used to select the optimal model using the largest value. ## The final value used for the model was mtry = 5. Выбираем mtry равным 4 и получаем Accuracy на тестовых данных. Замечу, что пришлось обучать модель на части от доступных тренировочных данных, т.к. для использования всех данных требуется больше оперативной памяти. Но, как показано в предыдущей работе, это не сильно повлияет на конечный результат.
library(randomForest) rfFit <- randomForest(label ~ ., data = train[sample(nrow(train), size = 15000),], mtry = 4) prediction_rf<-predict(rfFit,test) table(test$label, prediction_rf) ## prediction_rf ## 0 1 2 3 4 5 6 7 8 9 ## 0 1608 0 6 3 4 1 20 2 9 0 ## 1 0 1828 9 9 3 5 3 2 9 0 ## 2 12 9 1562 16 15 5 6 19 31 1 ## 3 12 1 26 1625 2 33 12 14 22 6 ## 4 0 6 11 1 1524 0 22 7 6 39 ## 5 12 3 3 48 12 1415 10 1 15 3 ## 6 13 4 8 0 4 11 1623 0 3 0 ## 7 3 14 25 2 13 3 0 1653 4 28 ## 8 4 10 12 64 8 21 12 5 1428 23 ## 9 4 4 10 39 38 10 0 39 7 1562 sum(diag(table(test$label, prediction_rf)))/nrow(test) ## [1] 0.9421989
SVM
И, наконец, Support Vector Machine. В этой модели будет использоваться Radial Kernel и подбираются уже два параметра: sigma (регуляризационный параметр) и C (параметр, определяющий форму ядра).
svmFit <- train(label ~ ., data = train_1000, method = "svmRadial", trControl = ctrl,tuneLength = 5) svmFit ## Support Vector Machines with Radial Basis Function Kernel ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 901, 900, 898, 900, 900, 901, ... ## Resampling results across tuning parameters: ## ## C Accuracy Kappa Accuracy SD Kappa SD ## 0.25 0.7862419 0.7612933 0.02209354 0.02469667 ## 0.50 0.8545924 0.8381166 0.02931921 0.03262332 ## 1.00 0.8826064 0.8694079 0.02903226 0.03225475 ## 2.00 0.8929180 0.8808766 0.02781461 0.03090255 ## 4.00 0.8986322 0.8872208 0.02607149 0.02898200 ## ## Tuning parameter 'sigma' was held constant at a value of 0.007650572 ## Accuracy was used to select the optimal model using the largest value. ## The final values used for the model were sigma = 0.007650572 and C = 4. grid <- expand.grid(C = 4:6, sigma = seq(0.006, 0.009, 0.001)) svmFit <- train(label ~ ., data = train_1000, method = "svmRadial", trControl = ctrl,tuneGrid=grid) svmFit ## Support Vector Machines with Radial Basis Function Kernel ## ## 1000 samples ## 70 predictor ## 10 classes: '0', '1', '2', '3', '4', '5', '6', '7', '8', '9' ## ## No pre-processing ## Resampling: Cross-Validated (10 fold, repeated 3 times) ## Summary of sample sizes: 901, 900, 900, 899, 901, 901, ... ## Resampling results across tuning parameters: ## ## C sigma Accuracy Kappa Accuracy SD Kappa SD ## 4 0.006 0.8943835 0.8824894 0.02999785 0.03335171 ## 4 0.007 0.8970537 0.8854531 0.02873482 0.03194698 ## 4 0.008 0.8984139 0.8869749 0.03068411 0.03410783 ## 4 0.009 0.8990838 0.8877269 0.03122154 0.03469947 ## 5 0.006 0.8960834 0.8843721 0.03061547 0.03404636 ## 5 0.007 0.8960703 0.8843617 0.03069610 0.03412880 ## 5 0.008 0.8990774 0.8877134 0.03083329 0.03427321 ## 5 0.009 0.8990838 0.8877271 0.03122154 0.03469983 ## 6 0.006 0.8957534 0.8840045 0.03094360 0.03441242 ## 6 0.007 0.8963971 0.8847267 0.03081294 0.03425451 ## 6 0.008 0.8990774 0.8877134 0.03083329 0.03427321 ## 6 0.009 0.8990838 0.8877271 0.03122154 0.03469983 ## ## Accuracy was used to select the optimal model using the largest value. ## The final values used for the model were sigma = 0.009 and C = 4. library(kernlab) svmFit <- ksvm(label ~ ., data = train,type="C-svc",kernel="rbfdot",kpar=list(sigma=0.008),C=4) prediction_svm <- predict(svmFit, newdata = test) table(test$label, prediction_svm) ## prediction_svm ## 0 1 2 3 4 5 6 7 8 9 ## 0 1625 0 5 1 0 3 13 0 6 0 ## 1 1 1841 6 6 4 1 0 3 5 1 ## 2 8 4 1624 5 7 1 1 13 11 2 ## 3 2 0 18 1684 0 23 2 6 12 6 ## 4 1 3 3 0 1567 0 9 7 5 21 ## 5 8 3 2 24 6 1465 7 0 6 1 ## 6 2 1 2 1 5 5 1649 0 1 0 ## 7 3 8 15 3 3 0 0 1695 3 15 ## 8 1 6 10 10 5 9 3 4 1530 9 ## 9 3 1 5 13 14 9 0 21 3 1644 sum(diag(table(test$label, prediction_svm)))/nrow(test) ## [1] 0.9717245
Ансамбль моделей
Создадим четвёртую модель, которая представляет собой ансамбль из трёх моделей, созданных ранее. Эта модель предсказывает то значение, за которое «голосует» большинство из использованных моделей.
all_prediction <- cbind(as.numeric(levels(prediction_knn))[prediction_knn], as.numeric(levels(prediction_rf))[prediction_rf], as.numeric(levels(prediction_svm))[prediction_svm]) predictions_ensemble <- apply(all_prediction, 1, function(row) { row %>% table(.) %>% which.max(.) %>% names(.) %>% as.numeric(.) }) table(test$label, predictions_ensemble) ## predictions_ensemble ## 0 1 2 3 4 5 6 7 8 9 ## 0 1636 0 5 1 0 1 8 0 2 0 ## 1 1 1851 3 5 3 0 0 1 4 0 ## 2 7 6 1636 3 6 0 0 11 7 0 ## 3 6 0 14 1690 1 18 4 8 7 5 ## 4 0 5 4 0 1573 0 12 6 2 14 ## 5 5 1 2 21 5 1478 7 0 3 0 ## 6 3 1 2 0 5 3 1651 0 1 0 ## 7 1 11 12 2 1 0 0 1704 0 14 ## 8 1 5 11 17 4 13 4 3 1514 15 ## 9 4 2 4 21 11 5 0 20 1 1645 sum(diag(table(test$label, predictions_ensemble)))/nrow(test) ## [1] 0.974939
Итоги
На тестовой выборке получены следующие результаты:
| Model | Test Accuracy |
| KNN | 0.981 |
| Random Forest | 0.948 |
| SVM | 0.971 |
| Ensemble | 0.974 |
Лучший показатель Accuracy имеет модель использующая метод k ближайших соседей (KNN).
Оценка моделей на сайте Kaggle приведена в следующей таблице.
| Model | Kaggle Accuracy |
| KNN | 0.97171 |
| Random Forest | 0.93286 |
| SVM | 0.97786 |
| Ensemble | 0.97471 |
И лучшие результаты здесь у SVM.
Eigenfaces
Ну и напоследок, уже из чистого любопытства, посмотрим наглядно на произведённые методом главных компонент преобразования. Для этого, во-первых получим изображение цифр в первоначальном виде.
set.seed(100) train_1000 <- data_train[sample(nrow(data_train), size = 1000),] colors<-c('white','black') cus_col<-colorRampPalette(colors=colors) default_par <- par() number_row <- 28 number_col <- 28 par(mfrow=c(5,5),pty='s',mar=c(1,1,1,1),xaxt='n',yaxt='n') for(i in 1:25) { z<-array(as.matrix(train_1000)[i,-1],dim=c(number_row,number_col)) z<-z[,number_col:1] image(1:number_row,1:number_col,z,main=train_1000[i,1],col=cus_col(256)) } par(default_par) 
И изображение этих же цифр, но уже после того, как мы использовали метод PCA и оставили первые 70 компонент. Получившиеся объекты принято называть eigenfaces
zero_var_col <- nearZeroVar(train_1000, saveMetrics = T) train_1000_cut <- train_1000[, !zero_var_col$nzv] pca <- prcomp(train_1000_cut[, -1], center = TRUE, scale = TRUE) restr <- pca$x[,1:70] %*% t(pca$rotation[,1:70]) restr <- scale(restr, center = FALSE , scale=1/pca$scale) restr <- scale(restr, center = -1 * pca$center, scale=FALSE) restr <- as.data.frame(cbind(train_1000_cut$label, restr)) test <- data.frame(matrix(NA, nrow = 1000, ncol = ncol(train_1000))) zero_col_number <- 1 for (i in 1:ncol(train_1000)) { if (zero_var_col$nzv[i] == F) { test[, i] <- restr[, zero_col_number] zero_col_number <- zero_col_number + 1 } else test[, i] <- train_1000[, i] } par(mfrow=c(5,5),pty='s',mar=c(1,1,1,1),xaxt='n',yaxt='n') for(i in 1:25) { z<-array(as.matrix(test)[i,-1],dim=c(number_row,number_col)) z<-z[,number_col:1] image(1:number_row,1:number_col,z,main=test[i,1],col=cus_col(256)) } par(default_par) 
В следующий раз мы рассмотрим одну из задач Text Mining’а, ну а пока можете присоединиться к курсу по Практическому анализу данных — рекомендую!
ссылка на оригинал статьи http://habrahabr.ru/post/267075/
Добавить комментарий