
Привет, Хабр!
Где-то месяц назад у меня появилось чувство постоянного беспокойства. Я стал плохо есть, еще хуже спать и постоянно читать-смотреть-слушать тонну новостей о пандемии. Исходя из них коронавирус то ли захватывал, то ли освобождал нашу планету, являлся то ли заговором масонов мировых правительств, а то ли местью панголина, вирус то ли угрожал всем и сразу, а то ли персонально мне и моему коту…
Сотни статей, постов в соцсетях, youtube-telegram-instagram-tik-tok (да уж простите) контента разной степени содержательности (и сомнительности) не приводили меня ни к чему, кроме уже описанного на Хабре разрушения мозга и еще большему чувству беспокойства.
Но в один день я купил гречки решил со всем этим нужно покончить. Как можно скорее!
Мне нужен был план действий чуть более осмысленный, чем тот, который у меня был до этого.
Все что мне нужно было это найти данные. Достоверные, полные, актуальные.
К моей радости это оказалось сделать значительно легче, чем представлялось мне, когда я осознал эту задачу.
Мой план:
- [+] найти достоверные, полные, актуальные данные о распространении COVID-19 в машиночитаемом формате, [1, 3]
- [+] читать статьи, в которых ни слова (почти) про COVID-19, а вместо этого описана «математика» распространения вирусных заболеваний,
- [+] участвовать в соревнованиях, где участники пытаются предсказать скорость распространения коронавируса [2],
- [+] сделать пару совершенно провальных попыток почитать релевантные исследования в области современно биологии и медицины,
- [-] перестать сраться в комментах к постам по COVID-19 в соцсетях,
- [+] собирать полученные знания в github репозиторий, открытый миру [3].
По прошествии месяца со дня формирования плана я выявил у него минимум один несомненный плюс – он оказался исполнимым. Но этот пост не о том, как пользоваться поисковой строкой для поиска нужного материала, а о том, что было дальше после реализации плана.
Вскоре я занялся анализом сложившейся ситуации. Ну как занялся – мне пришлось – стало ясно, что очень часто подходы, которые применяются меня не устраивали по ряду причин, о которых ниже.
Загружаем данные о распространении COVID-19:
#' #' Load COVID-19 spread: infected, recovered, and fatal cases #' Source: https://github.com/CSSEGISandData/COVID-19/tree/master/csse_covid_19_data/csse_covid_19_time_series #' load_covid_spread <- function() { require(dplyr) require(data.table) require(purrr) require(tidyr) load_time_series <- function(.case_type) { path_pattern <- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_%s_global.csv" fread(sprintf(path_pattern, .case_type)) %>% rename(country = `Country/Region`) %>% select(-c(`Province/State`, Lat, Long)) %>% group_by(country) %>% summarise_if(is.numeric, sum) %>% ungroup %>% gather(key = "date", value = "n", -country) %>% mutate(date = mdy(date)) } dt <- load_time_series("confirmed") %>% rename(confirmed_n = n) %>% inner_join( load_time_series("recovered") %>% rename(recovered_n = n), by = c("country", "date") ) %>% inner_join( load_time_series("deaths") %>% rename(deaths_n = n), by = c("country", "date") ) stopifnot(nrow(dt) > 0) return(dt) } spread_raw <- load_covid_spread() spread_raw %>% sample_n(10)
Загружаем данные о популяции стран:
#' #' Load countries stats #' Source: https://ods.ai/competitions/sberbank-covid19-forecast #' load_countries_stats <- function() { require(dplyr) require(magrittr) dt <- fread("https://raw.githubusercontent.com/codez0mb1e/covid-2019/master/data/countries.csv") dt %<>% select(-c(iso_alpha2, iso_numeric, name, official_name)) stopifnot(nrow(dt) > 0) return(dt) } countries_raw <- load_countries_stats() countries_raw %>% sample_n(10)
Предобработка данных:
data <- spread_raw %>% # add country population inner_join( countries_raw %>% transmute(ccse_name, country_iso = iso_alpha3, population) %>% filter(!is.na(country_iso)), by = c("country" = "ccse_name") ) %>% # calculate active cases mutate( active_n = confirmed_n - recovered_n - deaths_n ) %>% # calculate cases per 1M population mutate_at( vars(ends_with("_n")), list("per_1M" = ~ ./population*1e6) ) ## Calculte number of days since... get_date_since <- function(dt, .case_type, .n) { dt %>% group_by(country) %>% filter_at(vars(.case_type), ~ . > .n) %>% summarise(since_date = min(date)) } data %<>% inner_join( data %>% get_date_since("confirmed_n", 0) %>% rename(since_1st_confirmed_date = since_date), by = "country" ) %>% inner_join( data %>% get_date_since("confirmed_n_per_1M", 1) %>% rename(since_1_confirmed_per_1M_date = since_date), by = "country" ) %>% inner_join( data %>% get_date_since("deaths_n_per_1M", .1) %>% rename(since_dot1_deaths_per_1M_date = since_date), by = "country" ) %>% mutate_at( vars(starts_with("since_")), list("n_days" = ~ difftime(date, ., units = "days") %>% as.numeric) ) %>% mutate_at( vars(ends_with("n_days")), list(~ if_else(. > 0, ., NA_real_)) )
Настройки для графиков:
theme_set(theme_minimal()) lab_caption <- paste0( "Data source: Novel Coronavirus (COVID-19) Cases provided by Johns Hopkins University Center for Systems Science. \n", sprintf("Last updated: %s. ", format(max(data$date), '%d %B, %Y')), "Source code: github.com/codez0mb1e/covid-2019" ) filter_countries <- function(dt) dt %>% filter(country_iso %in% c("KOR", "ITA", "RUS", "CHN", "USA"))
Абсолютные числа
Проблема: абсолютное количество случаев заболевания как ключевой показатель.
Мотивация: если брать абсолютное количество заболевших, то деревня, в которой из 100 человек заболело 50, в сотни раз лучше Рима (к примеру) в смысле эпидемиологической обстановки.
Решение: отображать долю населения страны (города), которая имеет заболевание.
Проведем эксперимент: построим графики зависимости количества инфицированных по времени.
ggplot(data %>% filter_countries, aes(x = date)) + geom_col(aes(y = confirmed_n), alpha = .9) + scale_x_date(date_labels = "%d %b", date_breaks = "7 days") + facet_grid(country ~ .) + labs(x = "", y = "# of cases", title = "COVID-19 Spread (over time)", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

В странах, где не набралось большого количества заболевших по сравнению с США, невозможно разобрать ничего: ни роста, ни падения, ни уж в тем более, переломных моментов в пандемии населения страны. Прологарифмируем количество заболевших, в надежде увидеть что-то, но станет, как будто у всех около 100K заболеваний.
ggplot(data %>% filter_countries, aes(x = date)) + geom_col(aes(y = confirmed_n), alpha = .9) + scale_x_date(date_labels = "%d %b", date_breaks = "7 days") + scale_y_log10() + facet_grid(country ~ .) + labs(x = "", y = "# of cases", title = "COVID-19 Spread (over time)", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

Теперь то же самое, только с количеством забелевших на 1 миллион населения.
ggplot(data %>% filter_countries, aes(x = date)) + geom_col(aes(y = confirmed_n_per_1M), alpha = .9) + scale_x_date(date_labels = "%d %b", date_breaks = "7 days") + facet_grid(country ~ .) + labs(x = "", y = "# of cases per 1M", subtitle = "Infected cases per 1 million popultation", title = "COVID-19 Spread (over time)", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

И в логарифмической шкале:
ggplot(data %>% filter_countries, aes(x = date)) + geom_col(aes(y = confirmed_n_per_1M), alpha = .9) + scale_x_date(date_labels = "%d %b", date_breaks = "7 days") + scale_y_log10() + facet_grid(country ~ .) + labs(x = "", y = "# of cases per 1M", subtitle = "Infected cases per 1 million popultation", title = "COVID-19 Spread (over time)", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

Я думаю, не надо указывать насколько лучше стали видны «тихие» гавани и страны, где с эпидемиологической обстановкой не так спокойно.
Точка отсчета
Проблема: при сравнении эпидемиологической обстановки использовать дату обнаружения вируса в стране (или еще хуже в Китае) в качестве точки отсчета.
Мотивация: эффект низкой базы (был один заболевший, стало 3, рост +200%); пропускаем момент, когда эпидемия в стране приняла характер пандемии.
Решение: используем даты, когда >1 инфицированного на миллион населения страны, >0.1 погибшего на миллион населения, как точки отчета.
Аналогично посмотрим на динамику увеличения количества случаев заболевания, начиная с первого обнаруженного случая.
ggplot( data %>% filter_countries %>% filter(!is.na(since_1st_confirmed_date_n_days)), aes(x = since_1st_confirmed_date_n_days) ) + geom_col(aes(y = confirmed_n), alpha = .9) + scale_y_log10() + facet_grid(country ~ .) + labs(x = "# of days since 1st infected case", y = "# of cases", subtitle = "Infected cases since 1st infected case", title = "COVID-19 Spread", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

И динамику увеличения количества заболевших на 1 миллион населения, начиная с момента, когда у нас появился минимум 1 инфицированный на миллион населения.
ggplot( data %>% filter_countries %>% filter(!is.na(since_1_confirmed_per_1M_date_n_days)), aes(x = since_1_confirmed_per_1M_date_n_days) ) + geom_col(aes(y = confirmed_n_per_1M), alpha = .9) + scale_y_log10() + xlim(c(0, 55)) + facet_grid(country ~ .) + labs(x = "# of days since 1 infected cases per 1M", y = "# of cases per 1M", title = "COVID-19 Spread", subtitle = "Since 1 infected cases per 1 million popultation", caption = lab_caption) + theme(plot.caption = element_text(size = 8))

Китай и Южная Корея значительно присели, виден рост заболевших в России и тренды на «успокоение» ситуации в Италии и США.
Кого считаем
Проблема: для решения задачи «когда это все закончится» строим прогноз на количестве инфицированных.
Мотивация: выздоровевшие пациенты и умершие уже никого не заразят, никто из-за них уже не ляжет в больницу.
Решение: строим прогнозы на количестве активных случаев (инфицированные случаи минус сумма смертельных случаев и вылеченных).
Построим график, где отобразим случаи заражения, выздоровления, смертельные случаи и количество активных случаев (синяя линия).
plot_data <- data %>% filter_countries %>% filter(!is.na(since_1_confirmed_per_1M_date_n_days)) %>% mutate( confirmed_n_per_1M = confirmed_n_per_1M, recovered_n_per_1M = -recovered_n_per_1M, deaths_n_per_1M = -deaths_n_per_1M ) %>% select( country, since_1_confirmed_per_1M_date_n_days, ends_with("_n_per_1M") ) %>% gather( key = "case_state", value = "n_per_1M", -c(country, since_1_confirmed_per_1M_date_n_days, active_n_per_1M) ) ggplot(plot_data, aes(x = since_1_confirmed_per_1M_date_n_days)) + geom_col(aes(y = n_per_1M, fill = case_state), alpha = .9) + geom_line(aes(y = active_n_per_1M), color = "#0080FF", size = .25) + scale_fill_manual(element_blank(), labels = c("confirmed_n_per_1M" = "Infected cases", "recovered_n_per_1M" = "Recovered cases", "deaths_n_per_1M" = "Fatal cases"), values = c("confirmed_n_per_1M" = "grey", "recovered_n_per_1M" = "gold", "deaths_n_per_1M" = "black")) + xlim(c(0, 55)) + facet_grid(country ~ ., scales = "free") + labs(x = "# of days since 1 infected cases per 1M", y = "# of cases per 1M", title = "COVID-19 Spread by Countries", subtitle = "Active cases trend since 1 infected cases per 1 million popultation. \nBlue line - infected cases minus recovered and fatal.\nNegative values indicate recovered and fatal cases.", caption = lab_caption) + theme( legend.position = "top", plot.caption = element_text(size = 8) )
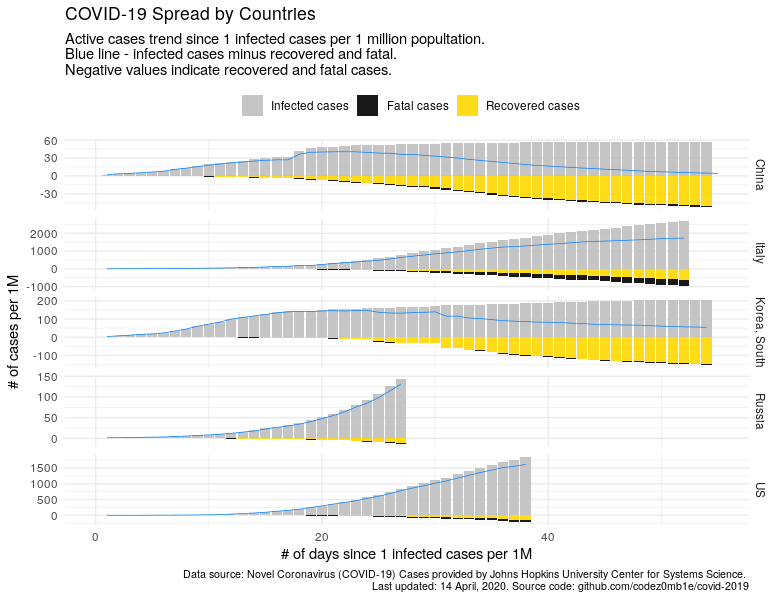
Из графика очевидно, насколько важна информация о количестве активных случаев для полного понимания развития эпидемиологической обстановки в соответствующей стане.
Все это было давно
Проблема: количество инфицированных случаев сегодня лишь фиксирование факта того, что было минимум неделю назад (скорее всего около 10 дней назад [я не знаю, как посчитать эту цифру]).
Решение: моделирование на данных недельной давности, проверка предсказания на сегодня; поиск инсайтов (например, отношение количества обнаруженных в день кейсов к количеству сделанных в этот день тестов). Простого решения, наверное, здесь нет.
Попробуем с помощью авторегрессионной модели ARIMA заглянуть на неделю вперед:
forecast_cases <- function(.country, .after_date, .forecast_horizont, .fun, ...) { dt <- data %>% # filter rows and cols filter( country == .country & date < .after_date ) %>% # convert to time-series arrange(date) %>% select(active_n_per_1M) dt %>% ts(frequency = 7) %>% # ARIMA model .fun(...) %>% # forecast forecast(h = .forecast_horizont) } forecast_horizont <- 7 after_date <- max(data$date) + days() countries_list <- c("Belgium", "France", "Italy", "Netherlands", "Norway", "Portugal", "Spain", "Switzerland", "US", "Russia", "China", "Korea, South") pred <- countries_list %>% map_dfr( function(.x) { m <- forecast_cases(.x, after_date, forecast_horizont, auto.arima) n_days_max <- max(data[data$country == .x, ]$since_1_confirmed_per_1M_date_n_days, na.rm = T) tibble( country = rep(.x, forecast_horizont), since_1_confirmed_per_1M_date_n_days = seq(n_days_max + 1, n_days_max + forecast_horizont, by = 1), pred = m$mean %>% as.numeric %>% round %>% as.integer, data_type = "Forecast" ) } ) plot_data <- data %>% filter(country %in% countries_list) %>% transmute( country, active_n_per_1M, since_1_confirmed_per_1M_date_n_days, data_type = "Historical data" ) %>% bind_rows( pred %>% rename(active_n_per_1M = pred) ) %>% mutate( double_every_14d = (1 + 1/14)^since_1_confirmed_per_1M_date_n_days, # double every 2 weeks double_every_7d = (1 + 1/7)^since_1_confirmed_per_1M_date_n_days, # double every week double_every_3d = (1 + 1/3)^since_1_confirmed_per_1M_date_n_days, # double every 3 days double_every_2d = (1 + 1/2)^since_1_confirmed_per_1M_date_n_days # double every 2 days ) active_n_per_1M_last <- plot_data %>% group_by(country) %>% arrange(desc(since_1_confirmed_per_1M_date_n_days)) %>% filter(row_number() == 1) %>% ungroup plot_data %<>% left_join( active_n_per_1M_last %>% transmute(country, active_n_per_1M_last = active_n_per_1M, since_1_confirmed_per_1M_date_n_days), by = c("country", "since_1_confirmed_per_1M_date_n_days") )
ggplot(plot_data, aes(x = since_1_confirmed_per_1M_date_n_days)) + geom_line(aes(y = double_every_7d), linetype = "dotted", color = "red", alpha = .65) + geom_line(aes(y = double_every_3d), linetype = "dotted", color = "red", alpha = .75) + geom_line(aes(y = double_every_2d), linetype = "dotted", color = "red", alpha = .85) + geom_line(aes(y = active_n_per_1M, color = country, linetype = data_type), show.legend = T) + geom_text(aes(y = active_n_per_1M_last + 20, label = country, color = country), hjust = 0.5, vjust = 0, check_overlap = T, show.legend = F, fontface = "bold", size = 3.6) + annotate(geom = "text", label = "Cases double \n every 2 days", x = 17, y = 1550, vjust = 0, size = 3.1) + annotate(geom = "text", label = "...every 3 days", x = 25, y = 1800, vjust = 0, size = 3.1) + annotate(geom = "text", label = "...every week", x = 50, y = 1500, vjust = 0, size = 3.1) + scale_linetype_manual(values = c("longdash", "solid")) + xlim(c(10, 55)) + ylim(c(0, max(plot_data$active_n_per_1M))) + labs(x = "# of days since 1 infected cases per 1M", y = "# of cases per 1M", title = "COVID-19 Spread by Countries", subtitle = "Active cases trend since 1 infected cases per 1 million popultation.", caption = lab_caption) + theme( legend.position = "bottom", legend.title = element_blank(), plot.caption = element_text(size = 8) )

Удивительный перелом в борьбе с COVID-19 демонстрирует Швейцария и Бельгия, в Португалии дела обстоят не сильно лучше, чем в США, у России есть плохой шанс расти быстрее, чем многие европейцы, которые показаны на графике.
Заключение
Но умаляю значения использования абсолютных величин количества заболевших, а также дат на графиках. Но для понимания динамики развития заболевания в конкретном регионе плотность активных заболевших, а также выбор правильной даты отсчета, является критичной.
Без учета размера популяции региона мы будем видеть просто страны с большим населением и пропускать локальные очаги вспышек инфекций. Без учета количества активных заболеваний (а не просто всех инфицированных) в топах возможно будут регионы, которые уже справились со вспышкой (как Китай). Неправильный выбор точки (даты) отсчета для сравнения эпидемиологической обстановки между странами также будет давать ложное преставление относительно течения заболеваний в сравниваемых регионах.
Воспроизвести результат, поиграться с другими странами, покопаться с исходниках, сделать pull-request можно здесь.
References
- COVID-19 Data Repository by Johns Hopkins CSSE, GitHub.
- Соревнования по прогнозированию: Kaggle COVID19 Global Forecasting, Sberbank COVID-19 Forecast.
- Код, материалы, первоисточники поста, GitHub.
Берегите себя!
ссылка на оригинал статьи https://habr.com/ru/post/497340/
Добавить комментарий